Die Abbildung ist ein Screenshot der interaktiven Karte der KEGG: Kyoto Encyclopedia of Genes and Genomes. An diesem Beispiel wird demonstriert, wie die KEGG-Datenbank aufgebaut ist. In Form eines INTERFACE zu ihr wird gezeigt, wie man einen vollständigen Zugang zu den Daten dieser Datenbank - d.h. zu allen am Stoffwechsel beteiligten Enzymen sowie den Intermediär- und Endprodukten erhalten kann
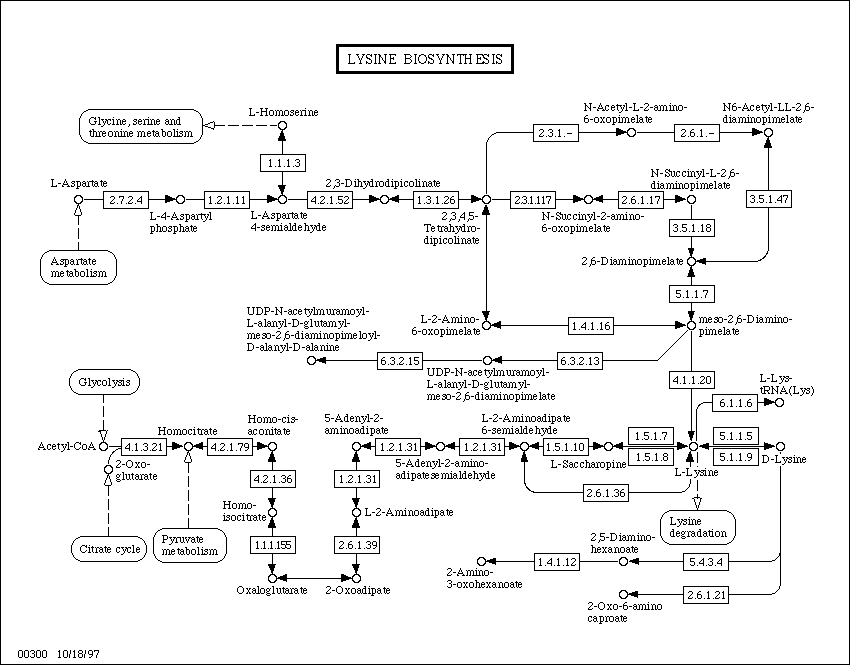
Dieser Kartenausschnitt enthält eine Vielzahl detaillierter Angaben. So enthält er die Namen der Verbindungen der Intermediär- und Stoffwechselendprodukte. Durch Anklicken des jeweiligen Kreises (Beispiel: Lysin) gelangt man zu einem Datenblatt, auf dem die Formel und die molekularen Eigenschaften der Verbindung aufgeführt sind. Ferner enthält das Datenblatt Angaben über Enzyme, die die jeweilige Verbindung umsetzen können.
Die biochemischen Reaktionen werden durch Enzyme katalysiert. Auf dem Kartenausschnitt sind sie als Ziffern wiedergegeben. Die Ziffern entstammen der EC-Nomenklatur. Durch Anklicken der entsprechenden Box gelangt man zu dem Datenblatt mit den Enzymangaben. Von dort wiederum gelangt man zu allen Daten, die für das jeweilige Enzym vorliegen. - Die Links auf den hier ausgewählten Datenblättern führen direkt zu den von KEGG vorgenommenen Eintragungen.
Die nachfolgenden Listen dienen dazu, einen systematischen Zugang zu allen Datenbankeintragungen von KEGG - Metabolic Pathways - zu erhalten (alle Links führen direkt zur Datenbank in Kyoto/Japan!)
| KEGG Metabolic PathwaysGraphical pathway maps and ortholog group tables |
|---|
Note - The ortholog group table contains only a subset of enzymes in the pathway map. The subset represents a correlated cluster of enzyme genes that appear in a localized area in the genome and that function at closely related positions in the reaction pathway.
Last updated: August 11, 1998
|
|